Forschungsgruppe Medizinische Systembiologie
Die Buchholz-Gruppe forscht auf den Gebieten der funktionellen Genomik und des Genome Editing. Unser Ziel ist es, krankheitsrelevante Gene zu identifizieren, funktionell zu charakterisieren und mit Hilfe verschiedener genetischer Werkzeuge (wie RNAi, CRISPR/Cas9 und Designer-Rekombinasen) zu korrigieren.
Genomsequenzierungen haben gezeigt, dass Krebsmutationen patientenspezifisch sind. Daher hat jedes Krebsgenom seinen individuellen genetischen Fingerabdruck. Das Buchholz-Labor entwickelt Technologien, um krebsauslösende Mutationen zu untersuchen und zu eliminieren. Hierdurch sollen individuelle Krebstherapien verbessert sowie die aktuellen Möglichkeiten der personalisierten Medizin vorangetrieben werden. Durch Ganzgenomsequenzierung ist es möglich, alle in einem Tumor vorhandenen Mutationen schnell zu identifizieren. Dennoch ist häufig nicht klar, welche dieser Mutationen tatsächlich für die Krankheit relevant sind. Die Entdeckung der RNA-Interferenz hat die Entwicklung von funktionellen Werkzeugen ermöglicht, die genomweite Loss-of-Function-Analysen in einem breiten Spektrum von Organismen erlauben. Somit ist es nun möglich, die Funktion jedes Gens in Zellen durch genomweite RNAi-Screens zu untersuchen. Darüber hinaus ist die CRISPR/Cas9 Technologie eines der revolutionärsten Werkzeuge in der Biotechnologie.
Unser Labor hat diese Technologie weiterentwickelt, um DNA-spezifisch an Stellen zu schneiden, die in Krebszellen mutiert sind. Darüber hinaus führen wir groß angelegte CRISPR-Screens in primärem Patientenmaterial durch, z. B. in Krebszelllinien und etablierten Krebsorganoiden, um Krebsmutationen funktionell zu untersuchen. Ein weiterer Schwerpunkt des Labors ist die Entwicklung von Genome Editing-Werkzeugen für die Präzisionsgenomchirurgie. Unter Anwendung modernster Technologien werden Designer-Rekombinasen und andere Genom-Editierwerkzeuge mit therapeutischem Potenzial entwickelt und sowohl in vitro als auch in vivo getestet.
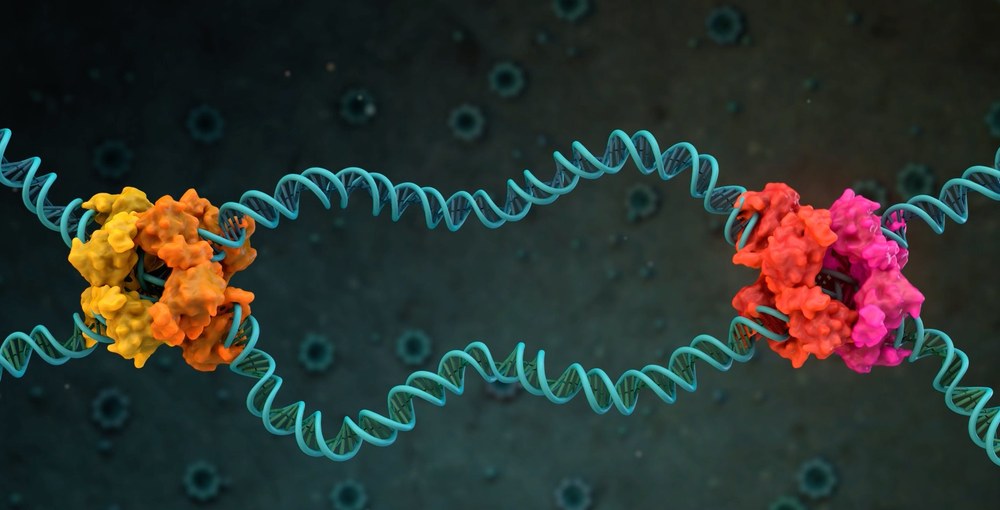
Modell einer Designer-Rekombinase, die eine krebsauslösende Mutation korrigiert. © Prof. Dr. Frank Buchholz, TU Dresden, Medizinische Fakultät
Methodenspektrum
- Sequenz-spezifische Rekombination
- Gezielte molekulare Evolution
- CRISPR/Cas9-Technologie
- Long-Read-PacBio-Sequenzierung
- Nanopore-Sequenzierung
- Hochdurchsatz- RNAi/CRISPR-Screens
- BAC-Tagging
- Live-Zell-Mikroskopie
- Organoid-Kultivierung
- Deep-Learning/KI
Kontakt
Prof. Dr. Frank Buchholz
Gruppenleiter Medizinische Systembiologie
Medizinische Fakultät Carl Gustav Carus, TU Dresden
Telefon: +49(0)351/463-40277
Email: frank.buchholz(at)tu-dresden.de
Sürün, D., Schneider, A., Mircetic, J., Neumann, K., Lansing, F., Paszkowski-Rogacz, M., Hänchen, V., Lee-Kirsch, M.A., Buchholz, F.
Efficient Generation and Correction of Mutations in Human iPS Cells Utilizing mRNAs of CRISPR Base Editors and Prime Editors.
Genes (Basel)May 6;11(5):511. doi: 10.3390/genes11050511. (2020)
Sayed, S., Paszkowski-Rogacz, M., Schmitt, L. T. and Buchholz, F.
CRISPR/Cas9 as a tool to dissect cancer mutations.
Methods 164-165: 36-48. (2019)
Camgoz, A., Paszkowski-Rogacz, M., Satpathy, S., Wermke, M., Hamann, M. V., von Bonin, M., Choudhary, C., Knapp, S. & Buchholz, F. (2018)
STK3 is a therapeutic target for a subset of acute myeloid leukemias.
Oncotarget, 9(39), 25458-25473. doi:10.18632/oncotarget.25238 (2018)
Mircetic, J., Dietrich, A., Paszkowski-Rogacz, M., Krause, M. & Buchholz, F.
Development of a genetic sensor that eliminates p53 deficient cells.
Nat Commun, 8(1), 1463. doi:10.1038/s41467-017-01688-w (2017)
Gebler, C., Lohoff, T., Paszkowski-Rogacz, M., Mircetic, J., Chakraborty, D., Camgoz, A., Hamann, M. V., Theis, M., Thiede, C. & Buchholz, F.
Inactivation of Cancer Mutations Utilizing CRISPR/Cas9.
J Natl Cancer Inst, 109(1). doi:10.1093/jnci/djw183 (2016)


