Forschungsgruppe Genetische Tumorrisikosyndrome
Jährlich wird bei Millionen von Menschen die Diagnose Krebs gestellt und tritt manchmal gehäuft bei mehreren Familienmitgliedern auf. Dies kann auf die Einwirkung gleicher Umweltfaktoren bzw. des gleichen Lebensstils zurückzuführen oder Zufall sein. Andererseits kann Krebs aber auch durch eine Erblichkeit bedingt sein, die zu einem höheren Risiko für Tumorerkrankungen führt und bei ca. 5-10 % aller Tumorerkrankungen vorliegt. Zusätzlich zum Auftreten der Tumorerkrankungen bei mehreren Familienmitgliedern sind genetische Tumorrisikosyndrome durch ein frühes Erkrankungsalter und eine höhere Wahrscheinlichkeit mehrerer Tumorerkrankungen bei einer Person charakterisiert. Eine genetische Testung kann die Ursache für ein Tumorrisikosyndrom in solchen Fällen aufdecken und spielt für die Beratung der Angehörigen und die Einschätzung ihres Tumorrisikos, die Empfehlung von Krebsvorsorgemaßnahmen oder prophylaktische Operationen und Therapieoptionen eine wichtige Rolle. Es gelingt jedoch nicht immer, mögliche genetische Ursachen bei Patienten mit der Fragestellung eines genetischen Tumorrisikosyndroms mit den aktuellen diagnostischen Methoden aufzudecken.
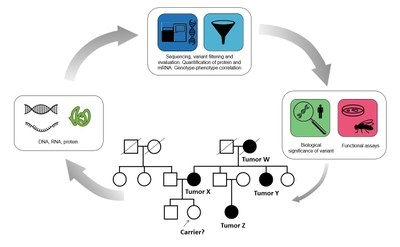
Abbildung 1: Um einen Träger einer genetischen Veränderung innerhalb einer Familie zu identifizieren, setzen wir Hochdurchsatztechnologien ein, um die Veränderungen in der DNA, der RNA und den Proteinen des Patienten zu ermitteln und sie mit einem Phänotyp zu korrelieren. Funktionelle und biologische Assays helfen uns, die Pathogenität der identifizierten Varianten zu verstehen. © A. Jahn /T.J. Widmann
„Genetische Tumorrisikosyndrome“ besser verstehen
Das Ziel der Forschungsgruppe ist es, unbekannte molekulare Ursachen für genetische Tumorrisikosyndrome aufzudecken. Versucht wird dies anhand von Untersuchungen der DNA sowie der aus der Erbinformation generierten RNA und der Proteine (Abbildung 1). Die komplexe Analyse von unterschiedlich breiten Charakterisierungen mittels Hochdurchsatz-Technologien (Omics) an gesunden sowie aus Tumorgewebe gewonnenen Patientenzellen ist Teil des diagnostischen Workflows des NCT/ DKTK-MASTER Programms, welches eine multizentrische Studie ist, die gezielte Therapieoptionen bei jung an Tumoren erkrankten Patient*innen oder Patient*innen mit seltenen Tumorerkrankungen untersucht. Innerhalb dieses Programms beurteilen wir genetische Varianten, die mit erblichen Tumorrisikosyndromen assoziiert sind und direkt klinische Konsequenzen haben können (A. Jahn et al., Ann. Oncol., 2022). Unser Ziel ist es, diese klinischen Konsequenzen der humangenetischen Beurteilung im Rahmen dieses Präzisionsonkologieprogramms besser zu verstehen bzw. die Informationsübertragung zu verbessern (Abbildung 2).

Abbildung 2: In einer genetischen Beratung informieren wir den Patienten und seine Familie darüber, wie sich genetische Veränderungen auf ihn und/oder seine Familie auswirken können. (c) Uniklinikum Dresden/Marc Eisele
Weiterhin analysieren wir in unabhängigen Forschungsprojekten zusätzlich die Pathogenität von Varianten unklarer Signifikanz in Tumorrisikogenen sowie Varianten in Kandidatengenen, die mit einer Erblichkeit assoziiert sein könnten. Wir charakterisieren die identifizierten genetischen Varianten und untersuchen die molekularen Signalwege, die in Patient*innen mit Tumorrisikosyndromen gestört sind. Langfristig könnten diese verschiedenen Herangehensweisen dazu führen, die diagnostischen und möglicherweise auch die therapeutischen Optionen für Patient*innen mit genetischen Tumorrisikosyndromen zu verbessern.
Angewandte Methoden
- DNA und RNA Sequenziermethoden (in Kooperation) und Omics-Datenanalyse
- Variantenbewertung für Tumorrisikogene und Kandidatengene
- Proteinbiochemische Methoden (Western Blot, Immunopräzipitation, Immunzytochemie, Reportergen-Assays)
- Transfektion, Knockdown und Überexpression, Gene-editing und funktionelle Analysen in Zellmodellen
Team
Postdoktorand
Tel.: +49 (0)351 458-11950
Email: AndresCruz.Garcia(at)uniklinikum-dresden.de
Wissenschaftliche Mitarbeiterin
Tel.: +49 (0)315 458-19598
Email: Alexandra.Baumann(at)uniklinikum-dresden.de
Frühere Mitglieder:
Johanna Hartig (Master Studentin)
Fathiya Nairoukh (Research Experience Program TU Dresden)
2022
Comprehensive cancer predisposition testing within the prospective MASTER trial identifies hereditary cancer patients and supports treatment decisions for rare cancers JahnA, RumpA, Widmann T J, Heining C, Horak P, Hutter B, Paramasivam N, Uhrig S, Gieldon L, Drukewitz S, Kübler A, Bermudez M, Hackmann K, Porrmann J, Wagner J, Arlt M, Franke M, Fischer J, Kowalzyk Z, WilliamD, Weth V, Oster S, Fröhlich M, Hüllein J, Orleś M, Valle González C, Kreutzfeldt S, Mock A, Heilig C E, Lipka D B, Möhrmann L, Hanf D, Teleanu V, Allgäuer M, Ruhnke L, Kutz O, Knurr A, Laßmann A, Endris V, Neumann O, Penzel R, Beck K, Richter D, Winter U, Wolf S, Pfütze , Geörg C, Meißburger B, Buchhalter I, Augustin M, Aulitzky W E, Hohenberger P, Kroiss M, Schirmacher P, Schlenk R F, Keilholz U, Klauschen F, Folprecht G, Bauer S, Siveke J T, Brandts C H, Kindler T, Boerries M, Illert A L, von Bubnoff N, Jost P J, Metzeler K H, Bitzer M, Schulze-Osthoff K, von Kalle C, Brors B, Stenzinger A, Weichert W, Hübschmann D, Fröhling S, Glimm H, Schröck E, Klink B. Ann Oncol. 2022 Aug 18;S0923-7534(22)01859-2. doi: 10.1016/j.annonc.2022.07.008
Comprehensive genomic and epigenomic analysis in cancer of unknown primary guides molecularly-informed therapies despite heterogeneity. Möhrmann L, Werner M, Oleś M, Mock A, Uhrig S, Jahn A, Kreutzfeldt S, Fröhlich M, Hutter B, Paramasivam N, Richter D, Beck K, Winter U, Pfütze K, Heilig CE, Teleanu V, Lipka DB, Zapatka M, Hanf D, List C, Allgäuer M, Penzel R, Rüter G, Jelas I, Hamacher R, Falkenhorst J, Wagner S, Brandts CH, Boerries M, Illert AL, Metzeler KH, Westphalen CB, Desuki A, Kindler T, Folprecht G, Weichert W, Brors B, Stenzinger A, Schröck E, Hübschmann D, Horak P, Heining C, Fröhling S, Glimm H. Nat Commun. 2022 Aug 2;13(1):4485. PMID: 35918329
2021
Comprehensive Genomic and Transcriptomic Analysis for Guiding Therapeutic Decisions in Patients with Rare Cancers. Horak P, Heining C, Kreutzfeldt S, Hutter B, Mock A, Hullein J, Frohlich M, Uhrig S, Jahn A, Rump A, Gieldon L, Mohrmann L, Hanf D, Teleanu V, Heilig CE, Lipka DB, Allgauer M, Ruhnke L, Lassmann A, Endris V, Neumann O, Penzel R, Beck K, Richter D, Winter U, Wolf S, Pfutze K, Georg C, Meissburger B, Buchhalter I, Augustin M, Aulitzky WE, Hohenberger P, Kroiss M, Schirmacher P, Schlenk RF, Keilholz U, Klauschen F, Folprecht G, Bauer S, Siveke JT, Brandts CH, Kindler T, Boerries M, Illert AL, von Bubnoff N, Jost PJ, Spiekermann K, Bitzer M, Schulze-Osthoff K, von Kalle C, Klink B, Brors B, Stenzinger A, Schrock E, Hubschmann D, Weichert W, Glimm H, Frohling S. Cancer Discov. 2021 Jun 10:candisc.0126.2021. doi: 10.1158/2159-8290.CD-21-0126. PMID: 34112699.
Recommendation and Acceptance of Counselling for Familial Cancer Risk in Newly Diagnosed Breast Cancer Cases. Kast K, Häfner J, Schröck E, Jahn A, Werner C, Meisel C, Wimberger P. Breast Care 2021. doi: 10.1159/000517021
Biallelic UBE4A loss-of-function variants cause intellectual disability and global developmental delay. Souto Melo U, Bonner D, Kent Lloyd K C...Di Donato N, Jahn A, Chedrawi A, Alkuraya F S, Kok F, Byers H M. Genet Med. 2021 Jan 8. PMID: 33420346
2020
The landscape of chromothripsis across adult cancer types. Voronina N, Wong JKL...Jahn A, ...Lichter P, Fröhling S, Ernst A. Nat Commun. 2020 May 8;11(1):2320. PMID: 32385320
Metastatic adult pancreatoblastoma: Multimodal treatment and molecular characterization of a very rare disease. Berger AK, Mughal SS, Allgäuer M, ... Jahn A, Schröck E, ...Stenzinger A, Fröhling S, Glimm H, Heining C. Pancreatology. 2020 Apr;20(3):425-432. PMID: 32156527
2019
Optimizing Genetic Workup in Pheochromocytoma and Paraganglioma by Integrating Diagnostic and Research Approaches. Gieldon L, William D, Hackmann K, Jahn W, Jahn A, Wagner J, ... Schröck E, Eisenhofer G, Richter S, Klink B. Cancers (Basel). 2019 Jun 11;11(6). PMID: 31212687
2018
Novel VPS33B mutation in a patient with autosomal recessive keratoderma-ichthyosis-deafness (ARKID) syndrome. Alter S, Hotz A, Jahn A, Di Donato N, ... Fischer J, Tzschach A.Am J Med Genet A. 2018 Dec;176(12):2862-2866. PMID: 30561130
Diagnostic value of partial exome sequencing in developmental disorders. Gieldon L, Mackenroth L, Kahlert AK, ... Jahn A, Kuhlee F, Hackmann K, Schrock E, Di Donato N, Rump A. PLoS One. 2018 Aug 9;13(8):e0201041. PMID: 30091983
Novel truncating PPM1D mutation in a patient with intellectual disability. Porrmann J, Rump A, Hackmann K, Di Donato N, Kahlert AK, Wagner J, Jahn A, Eger I, Flury M, Schrock E, Tzschach A, Gieldon L. Eur J Med Genet. 2018 May 11. pii: S1769-7212(18)30067-3. PMID: 29758292
2017
ZBTB48 is both a vertebrate telomere-binding protein and a transcriptional activator. Jahn A*, Rane G*, Paszkowski-Rogacz M, Sayols S, Bluhm A, Han CT, Draškovič I, Londoño-Vallejo JA, Kumar AP, Buchholz F, Butter F, Kappei D. EMBO Rep. 2017 Jun;18(6):929-946. PMID: 28500257 *These authors contributed equally to this work